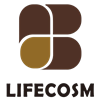
Produtos
Kit de detecção Lifecosm SARS-Cov-2-RT-PCR para 2019-nCoV
Uso esperado
Este kit é usado para a detecção qualitativa do novo coronavírus (2019-nCoV) usando swabs de garganta, swabs nasofaríngeos, fluido de lavagem broncoalveolar e escarro. O resultado da detecção deste produto é apenas para referência clínica e não deve ser usado como única evidência para diagnóstico e tratamento clínico. Uma análise abrangente da condição é recomendada em combinação com as manifestações clínicas do paciente e outros exames laboratoriais.
Princípio de inspeção
O kit baseia-se na tecnologia RT-PCR de uma etapa. De fato, os genes ORF1ab e N do novo coronavírus de 2019 (2019-nCoV) foram selecionados como regiões-alvo de amplificação. Primers específicos e sondas fluorescentes (as sondas do gene N são marcadas com FAM e as sondas do ORF1ab são marcadas com HEX) foram projetados para detectar o RNA do novo coronavírus de 2019 em amostras. O kit também inclui um sistema de detecção de controle interno endógeno (sonda do gene de controle interno marcada com CY5) para monitorar o processo de coleta da amostra e amplificação do RNA e da PCR, reduzindo assim os resultados falso-negativos.
Componentes principais
| Componentes | Volume(48T/Kit) |
| Solução de reação RT-PCR | 96µl |
| Mistura de sonda TaqMan do iniciador nCOV (ORF1ab, gene N, gene RnaseP) | 864µl |
| Controle negativo | 1500µl |
| Controle positivo nCOV (gene ORF1ab N) | 1500µl |
Reagentes próprios: reagentes de extração ou purificação de RNA. Controle negativo/positivo: O controle positivo é o RNA contendo o fragmento alvo, enquanto o controle negativo é água sem ácido nucleico. Durante o uso, eles devem participar da extração e devem ser considerados infecciosos. Devem ser manuseados e descartados de acordo com as normas aplicáveis.
O gene de referência interno é o gene humano RnaseP.
Condições de armazenamento e data de validade
-20±5℃, evite congelamento e descongelamento repetidos mais de 5 vezes, válido por 6 meses.
Instrumento aplicável
Com FAM / HEX / CY5 e outros instrumentos de PCR fluorescentes multicanal.
Requisitos de amostra
1. Tipos de espécimes aplicáveis: swabs de garganta, swabs nasofaríngeos, fluido de lavagem broncoalveolar, escarro.
2.Coleta de amostras (técnica asséptica)
Cotonete faríngeo: limpe as amígdalas e a parede posterior da faringe com dois cotonetes ao mesmo tempo e, em seguida, mergulhe a cabeça do cotonete em um tubo de ensaio contendo a solução de amostragem
Escarro: Após o paciente tossir profundamente, coletar o escarro tossido em um tubo de ensaio com tampa de rosca contendo a solução de amostragem; fluido de lavagem broncoalveolar: Amostragem por profissionais médicos. 3. Armazenamento e transporte de amostras
As amostras para isolamento viral e teste de RNA devem ser testadas o mais rápido possível. As amostras que podem ser detectadas em até 24 horas podem ser armazenadas a 4°C; aquelas que não podem ser detectadas em até 24 horas podem ser armazenadas a 4°C.
horas devem ser armazenadas a -70 ℃ ou menos (se não houver condição de armazenamento de -70 ℃, elas devem ser
(armazenado temporariamente a -20°C em geladeira). As amostras devem evitar congelamento e descongelamento repetidos durante o transporte. As amostras devem ser enviadas ao laboratório o mais rápido possível após a coleta. Se as amostras precisarem ser transportadas por longas distâncias, recomenda-se o armazenamento em gelo seco.
Métodos de teste
1 Processamento de amostras e extração de RNA (área de processamento de amostras)
Recomenda-se coletar 200 μl de amostra líquida para extração de RNA. Para as etapas de extração relacionadas, consulte as instruções dos kits comerciais de extração de RNA. Tanto o negativo quanto o negativo
os controles neste kit estavam envolvidos na extração.
2 Preparação de reagentes de PCR (área de preparação de reagentes)
2.1 Remova todos os componentes do kit, descongele e misture à temperatura ambiente. Centrifugue a 8.000 rpm por alguns segundos antes de usar; calcule a quantidade necessária de reagentes e prepare o sistema de reação conforme mostrado na tabela a seguir:
| Componentes | Porção N (sistema de 25µl) |
| Mistura de sonda TaqMan do iniciador nCOV | 18 µl × N |
| Solução de reação RT-PCR | 2 µl × N |
| *N = número de amostras testadas + 1 (controle negativo) + 1 (nCOVcontrole positivo) | |
2.2 Após misturar bem os componentes, centrifugue por um curto período para deixar todo o líquido na parede do tubo cair no fundo do tubo e, em seguida, aliquote 20 µl do sistema de amplificação no tubo de PCR.
3 Amostragem (área de preparação de amostras)
Adicione 5 μl dos controles negativo e positivo após a extração. O RNA da amostra a ser testada é adicionado ao tubo de reação de PCR.
Tampe bem o tubo e centrifugue a 8.000 rpm por alguns segundos antes de transferi-lo para a área de detecção de amplificação.
4 Amplificação por PCR (área de detecção amplificada)
4.1 Coloque o tubo de reação na célula de amostra do instrumento e defina os parâmetros da seguinte forma:
| estágio | Ciclo número | Temperatura(°C) | Tempo | coleçãosite |
| Revertertranscrição | 1 | 42 | 10 minutos | - |
| Pré-desnaturaçãon | 1 | 95 | 1 minuto | - |
| Ciclo | 45 | 95 | 15s | - |
| 60 | 30 anos | coleta de dados |
Seleção do canal de detecção do instrumento: Selecione o canal FAM, HEX e CY5 para o sinal de fluorescência. Para fluorescência de referência, NENHUM, não selecione ROX.
5 Análise de resultados (consulte as instruções experimentais de cada instrumento para configuração)
Após a reação, salve os resultados. Após a análise, ajuste o valor inicial, o valor final e o valor limite da linha de base de acordo com a imagem (o usuário pode ajustar de acordo com a situação real, o valor inicial pode ser definido como 3 a 15, o valor final pode ser definido como 5 a 20, ajuste) no gráfico logarítmico. No limite da janela, a linha limite está na fase logarítmica e a curva de amplificação do controle negativo é uma linha reta ou abaixo da linha limite.
6 Controle de qualidade (um controle de procedimento está incluído no teste) Controle negativo: nenhuma curva de amplificação óbvia para canais de detecção FAM, HEX e CY5
Controle positivo de COV: curva de amplificação óbvia dos canais de detecção FAM e HEX, valor Ct ≤ 32, mas nenhuma curva de amplificação do canal CY5;
Os requisitos acima devem ser atendidos simultaneamente no mesmo experimento; caso contrário, o experimento será inválido e precisará ser repetido.
7 Determinação dos resultados.
7.1 Se não houver curva de amplificação ou valor de Ct > 40 nos canais FAM e HEX da amostra de teste, e houver uma curva de amplificação no canal CY5, pode-se concluir que não há RNA do novo coronavírus de 2019 (2019-nCoV) na amostra;
.2 Se a amostra de teste apresentar curvas de amplificação óbvias nos canais FAM e HEX, e o valor de Ct for ≤40, pode-se considerar que a amostra é positiva para o novo coronavírus de 2019 (2019-nCoV).
7.3 Se a amostra de teste apresentar uma curva de amplificação nítida apenas em um canal de FAM ou HEX, e o valor de Ct for ≤ 40, e não houver curva de amplificação no outro canal, os resultados precisam ser testados novamente. Se os resultados do novo teste forem consistentes, a amostra pode ser considerada positiva para o novo canal.
coronavírus 2019 (2019-nCoV). Se o resultado do novo teste for negativo, pode-se concluir que a amostra é negativa para o novo coronavírus de 2019 (2019-nCoV).
Valor de julgamento positivo
O método da curva ROC é usado para determinar o valor de referência do TC do kit e o valor de referência do controle interno é 40.
Interpretação dos resultados dos testes
1. Cada experimento deve ser testado para controles negativos e positivos. Os resultados dos testes só podem ser determinados quando os controles atendem aos requisitos de controle de qualidade.
2. Quando os canais de detecção FAM e HEX são positivos, o resultado do canal CY5 (canal de controle interno) pode ser negativo devido à competição do sistema.
3. Quando o resultado do controle interno for negativo, se os canais de detecção FAM e HEX do tubo de ensaio também forem negativos, significa que o sistema está desabilitado ou a operação está incorreta, o teste é inválido. Portanto, as amostras precisam ser testadas novamente.